IAMB 알고리즘을 사용한 기능 선택: 기계 학습에 대한 간단한 다이빙
자, 이야기는 이렇습니다. 저는 최근 Zhuang 교수의 학교 과제에서 Incremental Association Markov Blanket(IAMB)라는 매우 멋진 알고리즘을 사용하는 작업을 수행했습니다. 저는 데이터 과학이나 통계에 대한 배경 지식이 없기 때문에 이것은 저에게 새로운 영역이지만 새로운 것을 배우는 것을 좋아합니다. 목표는? IAMB를 사용하여 데이터세트에서 기능을 선택하고 이것이 머신러닝 모델의 성능에 어떤 영향을 미치는지 확인하세요.
IAMB 알고리즘의 기본 사항을 살펴보고 이를 Jason Brownlee의 데이터 세트의 Pima Indians Diabetes 데이터 세트에 적용하겠습니다. 이 데이터 세트는 여성의 건강 데이터를 추적하고 당뇨병 여부를 포함합니다. 우리는 IAMB를 사용하여 당뇨병 예측에 가장 중요한 특징(BMI 또는 혈당 수치 등)을 알아낼 것입니다.
IAMB 알고리즘은 무엇이며 왜 사용합니까?
IAMB 알고리즘은 수수께끼 속의 용의자 목록을 정리하는 데 도움을 주는 친구와 같습니다. 목표를 예측하는 데 정말로 중요한 변수만 골라내도록 설계된 특징 선택 방법입니다. 이 경우 대상은 당뇨병 여부입니다.
- 전진 단계: 대상과 밀접한 관련이 있는 변수를 추가합니다.
- 후진 단계: 실제로 도움이 되지 않는 변수를 잘라내고 가장 중요한 변수만 남도록 합니다.
간단히 말하면 IAMB는 가장 관련성이 높은 기능만 선택하여 데이터 세트가 복잡해지는 것을 방지하는 데 도움이 됩니다. 이는 모델 성능을 단순하게 유지하고 훈련 시간을 단축하려는 경우 특히 유용합니다.
출처: 대규모 마르코프 담요 발견을 위한 알고리즘
이 알파 기능은 무엇이며 왜 중요한가요?
여기서 알파가 필요합니다. 통계에서 알파(α)는 "통계적으로 유의미한" 항목을 결정하기 위해 설정한 임계값입니다. 교수가 제공한 지침의 일부로 알파 0.05를 사용했습니다. 즉, 대상 변수와 무작위로 연관될 확률이 5% 미만인 특성만 유지하고 싶다는 뜻입니다. 따라서 특성의 p-값이 0.05보다 작다면 이는 목표와 강력하고 통계적으로 유의미한 연관성이 있음을 의미합니다.
이 알파 임계값을 사용하여 가장 의미 있는 변수에만 초점을 맞추고 "유의성" 테스트를 통과하지 못한 변수는 무시합니다. 가장 관련성이 높은 기능은 유지하고 노이즈는 걸러내는 필터와 같습니다.
실습: Pima Indians 당뇨병 데이터 세트에서 IAMB 사용
설정은 다음과 같습니다. Pima Indians 당뇨병 데이터 세트에는 건강 기능(혈압, 연령, 인슐린 수치 등)과 목표 결과(당뇨병 발병 여부)가 있습니다.
먼저 데이터를 로드하고 확인합니다.
import pandas as pd # Load and preview the dataset url = 'https://raw.githubusercontent.com/jbrownlee/Datasets/master/pima-indians-diabetes.data.csv' column_names = ['Pregnancies', 'Glucose', 'BloodPressure', 'SkinThickness', 'Insulin', 'BMI', 'DiabetesPedigreeFunction', 'Age', 'Outcome'] data = pd.read_csv(url, names=column_names) print(data.head())
Alpha = 0.05로 IAMB 구현
다음은 IAMB 알고리즘의 업데이트된 버전입니다. 우리는 p-값을 사용하여 유지할 특성을 결정하므로 p-값이 알파(0.05)보다 작은 특성만 선택됩니다.
import pingouin as pg
def iamb(target, data, alpha=0.05):
markov_blanket = set()
# Forward Phase: Add features with a p-value alpha
for feature in list(markov_blanket):
reduced_mb = markov_blanket - {feature}
result = pg.partial_corr(data=data, x=feature, y=target, covar=reduced_mb)
p_value = result.at[0, 'p-val']
if p_value > alpha:
markov_blanket.remove(feature)
return list(markov_blanket)
# Apply the updated IAMB function on the Pima dataset
selected_features = iamb('Outcome', data, alpha=0.05)
print("Selected Features:", selected_features)
이 프로그램을 실행했을 때 IAMB가 당뇨병 결과와 가장 밀접하게 관련되어 있다고 생각하는 기능의 세련된 목록이 제공되었습니다. 이 목록은 모델을 구축하는 데 필요한 변수의 범위를 좁히는 데 도움이 됩니다.
Selected Features: ['BMI', 'DiabetesPedigreeFunction', 'Pregnancies', 'Glucose']
IAMB가 선택한 기능이 모델 성능에 미치는 영향 테스트
선택한 기능이 있으면 실제 테스트에서는 모든 기능과 IAMB에서 선택한 기능의 모델 성능을 비교합니다. 이를 위해 간단한 Gaussian Naive Bayes 모델을 사용했습니다. 왜냐하면 이 모델은 간단하고 (전체 베이지안 분위기와 관련이 있는) 확률과 잘 어울리기 때문입니다.
모델을 학습하고 테스트하는 코드는 다음과 같습니다.
from sklearn.model_selection import train_test_split
from sklearn.naive_bayes import GaussianNB
from sklearn.metrics import accuracy_score, f1_score, roc_auc_score
# Split data
X = data.drop('Outcome', axis=1)
y = data['Outcome']
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.3, random_state=42)
# Model with All Features
model_all = GaussianNB()
model_all.fit(X_train, y_train)
y_pred_all = model_all.predict(X_test)
# Model with IAMB-Selected Features
X_train_selected = X_train[selected_features]
X_test_selected = X_test[selected_features]
model_iamb = GaussianNB()
model_iamb.fit(X_train_selected, y_train)
y_pred_iamb = model_iamb.predict(X_test_selected)
# Evaluate models
results = {
'Model': ['All Features', 'IAMB-Selected Features'],
'Accuracy': [accuracy_score(y_test, y_pred_all), accuracy_score(y_test, y_pred_iamb)],
'F1 Score': [f1_score(y_test, y_pred_all, average='weighted'), f1_score(y_test, y_pred_iamb, average='weighted')],
'AUC-ROC': [roc_auc_score(y_test, y_pred_all), roc_auc_score(y_test, y_pred_iamb)]
}
results_df = pd.DataFrame(results)
display(results_df)
결과
비교 내용은 다음과 같습니다.
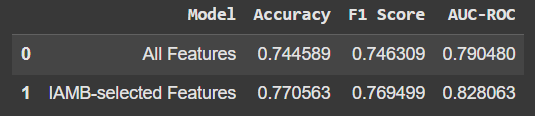
IAMB가 선택한 기능만 사용하면 정확도와 기타 측정항목이 약간 향상되었습니다. 큰 발전은 아니지만 더 적은 수의 기능으로 더 나은 성능을 얻을 수 있다는 사실은 유망합니다. 게다가 이는 우리 모델이 '노이즈'나 관련 없는 데이터에 의존하지 않는다는 것을 의미합니다.
주요 시사점
- IAMB는 기능 선택에 적합합니다: 목표를 예측하는 데 정말로 중요한 것에만 집중하여 데이터 세트를 정리하는 데 도움이 됩니다.
- 적을수록 더 많은 경우가 많습니다: 여기서 본 것처럼 모델 정확도가 약간 향상되는 것처럼 특성이 적을수록 더 나은 결과를 얻을 수도 있습니다.
- 학습과 실험은 재미있는 부분입니다: 데이터 과학에 대한 깊은 배경 지식이 없더라도 이와 같은 프로젝트에 참여하면 데이터와 기계 학습을 이해하는 새로운 방법이 열립니다.
이것이 IAMB에 대한 친근한 소개가 되기를 바랍니다! 궁금하다면 한번 시도해 보세요. 기계 학습 도구 상자에 있는 편리한 도구이며 자신의 프로젝트에서 몇 가지 멋진 개선 사항을 확인할 수도 있습니다.
출처: 대규모 마르코프 담요 발견을 위한 알고리즘
-
 MySQL을 사용하여 오늘 생일을 가진 사용자를 어떻게 찾을 수 있습니까?MySQL을 사용하여 오늘 생일이 있는 사용자를 식별하는 방법MySQL을 사용하여 오늘이 사용자의 생일인지 확인하려면 생일이 일치하는 모든 행을 찾는 것이 필요합니다. 오늘 날짜. 이는 UNIX 타임스탬프로 저장된 생일을 오늘 날짜와 비교하는 간단한 MySQL 쿼리를 ...프로그램 작성 2024년 11월 18일에 게시됨
MySQL을 사용하여 오늘 생일을 가진 사용자를 어떻게 찾을 수 있습니까?MySQL을 사용하여 오늘 생일이 있는 사용자를 식별하는 방법MySQL을 사용하여 오늘이 사용자의 생일인지 확인하려면 생일이 일치하는 모든 행을 찾는 것이 필요합니다. 오늘 날짜. 이는 UNIX 타임스탬프로 저장된 생일을 오늘 날짜와 비교하는 간단한 MySQL 쿼리를 ...프로그램 작성 2024년 11월 18일에 게시됨 -
 단위가 없는 CSS 변수를 다른 단위와 함께 사용하려면 어떻게 해야 합니까?유연성 있게 단위 없는 CSS 변수를 사용하는 방법단위 없는 CSS 변수는 스타일시트 전체에서 편리하게 사용할 수 있는 숫자 값을 저장하는 기능을 제공합니다. . 그러나 백분율이나 픽셀과 같은 다양한 단위가 필요한 다양한 컨텍스트에서 동일한 변수를 사용하려는 시나리오가...프로그램 작성 2024년 11월 18일에 게시됨
단위가 없는 CSS 변수를 다른 단위와 함께 사용하려면 어떻게 해야 합니까?유연성 있게 단위 없는 CSS 변수를 사용하는 방법단위 없는 CSS 변수는 스타일시트 전체에서 편리하게 사용할 수 있는 숫자 값을 저장하는 기능을 제공합니다. . 그러나 백분율이나 픽셀과 같은 다양한 단위가 필요한 다양한 컨텍스트에서 동일한 변수를 사용하려는 시나리오가...프로그램 작성 2024년 11월 18일에 게시됨 -
 Svelte(Kit)에서 #await 블록이 해결될 때 함수 실행콘텐츠로 건너뛰기: svelte의 #await 블록 정보 #await 블록이 해결되거나 거부될 때 함수 실행(트리거) 브라우저에 표시되는 정의되지 않은 텍스트 또는 반환된 텍스트 수정 1. 방법 1(빈 문자열 반환): 2. 방법 2(CSS를 사용하여 UI의 함수에서 반...프로그램 작성 2024년 11월 18일에 게시됨
Svelte(Kit)에서 #await 블록이 해결될 때 함수 실행콘텐츠로 건너뛰기: svelte의 #await 블록 정보 #await 블록이 해결되거나 거부될 때 함수 실행(트리거) 브라우저에 표시되는 정의되지 않은 텍스트 또는 반환된 텍스트 수정 1. 방법 1(빈 문자열 반환): 2. 방법 2(CSS를 사용하여 UI의 함수에서 반...프로그램 작성 2024년 11월 18일에 게시됨 -
 `if` 문 너머: 명시적인 `bool` 변환이 있는 유형을 형변환 없이 사용할 수 있는 다른 곳은 어디입니까?형변환 없이 허용되는 bool로의 상황별 변환귀하의 클래스는 bool로의 명시적인 변환을 정의하여 해당 인스턴스 't'를 조건문에서 직접 사용할 수 있도록 합니다. 그러나 이 명시적인 변환은 다음과 같은 질문을 제기합니다. 캐스트 없이 't'...프로그램 작성 2024년 11월 18일에 게시됨
`if` 문 너머: 명시적인 `bool` 변환이 있는 유형을 형변환 없이 사용할 수 있는 다른 곳은 어디입니까?형변환 없이 허용되는 bool로의 상황별 변환귀하의 클래스는 bool로의 명시적인 변환을 정의하여 해당 인스턴스 't'를 조건문에서 직접 사용할 수 있도록 합니다. 그러나 이 명시적인 변환은 다음과 같은 질문을 제기합니다. 캐스트 없이 't'...프로그램 작성 2024년 11월 18일에 게시됨 -
 단일 Java 파일에 여러 클래스를 가질 수 있습니까?Java 파일의 여러 클래스Java에서는 단일 .java 파일 내에 여러 클래스를 가질 수 있습니다. 단, Public 최상위 클래스는 하나만 있을 수 있으며 소스 파일과 이름이 동일해야 합니다.파일에 여러 클래스를 두는 목적은 논리적으로 관련된 코드를 구성하는 것입니...프로그램 작성 2024년 11월 18일에 게시됨
단일 Java 파일에 여러 클래스를 가질 수 있습니까?Java 파일의 여러 클래스Java에서는 단일 .java 파일 내에 여러 클래스를 가질 수 있습니다. 단, Public 최상위 클래스는 하나만 있을 수 있으며 소스 파일과 이름이 동일해야 합니다.파일에 여러 클래스를 두는 목적은 논리적으로 관련된 코드를 구성하는 것입니...프로그램 작성 2024년 11월 18일에 게시됨 -
 Bootstrap 4 Beta의 열 오프셋은 어떻게 되었나요?Bootstrap 4 베타: 열 오프셋 제거 및 복원Bootstrap 4는 베타 1 릴리스에서 열 오프셋 방식에 중요한 변경 사항을 도입했습니다. 열이 오프셋되었습니다. 그러나 후속 베타 2 릴리스에서는 이러한 변경 사항이 취소되었습니다.offset-md-*에서 ml-...프로그램 작성 2024년 11월 18일에 게시됨
Bootstrap 4 Beta의 열 오프셋은 어떻게 되었나요?Bootstrap 4 베타: 열 오프셋 제거 및 복원Bootstrap 4는 베타 1 릴리스에서 열 오프셋 방식에 중요한 변경 사항을 도입했습니다. 열이 오프셋되었습니다. 그러나 후속 베타 2 릴리스에서는 이러한 변경 사항이 취소되었습니다.offset-md-*에서 ml-...프로그램 작성 2024년 11월 18일에 게시됨 -
 고유 ID를 유지하고 중복 이름을 처리하면서 PHP에서 두 개의 연관 배열을 어떻게 결합합니까?PHP에서 연관 배열 결합PHP에서는 두 개의 연관 배열을 단일 배열로 결합하는 것이 일반적인 작업입니다. 다음 요청을 고려하십시오.문제 설명:제공된 코드는 두 개의 연관 배열 $array1 및 $array2를 정의합니다. 목표는 두 배열의 모든 키-값 쌍을 통합하는 ...프로그램 작성 2024년 11월 18일에 게시됨
고유 ID를 유지하고 중복 이름을 처리하면서 PHP에서 두 개의 연관 배열을 어떻게 결합합니까?PHP에서 연관 배열 결합PHP에서는 두 개의 연관 배열을 단일 배열로 결합하는 것이 일반적인 작업입니다. 다음 요청을 고려하십시오.문제 설명:제공된 코드는 두 개의 연관 배열 $array1 및 $array2를 정의합니다. 목표는 두 배열의 모든 키-값 쌍을 통합하는 ...프로그램 작성 2024년 11월 18일에 게시됨 -
 PDO 데이터베이스 연결을 테스트하고 오류를 효과적으로 처리하는 방법은 무엇입니까?PDO 데이터베이스 연결 테스트데이터베이스 설치를 개발할 때 데이터베이스 연결의 유효성을 확인하는 것이 중요합니다. 이는 기본 설정을 구성하려고 할 때 특히 중요합니다. PDO(PHP 데이터 개체)는 유효한 연결과 유효하지 않은 연결을 모두 테스트하는 효율적인 방법을 ...프로그램 작성 2024년 11월 18일에 게시됨
PDO 데이터베이스 연결을 테스트하고 오류를 효과적으로 처리하는 방법은 무엇입니까?PDO 데이터베이스 연결 테스트데이터베이스 설치를 개발할 때 데이터베이스 연결의 유효성을 확인하는 것이 중요합니다. 이는 기본 설정을 구성하려고 할 때 특히 중요합니다. PDO(PHP 데이터 개체)는 유효한 연결과 유효하지 않은 연결을 모두 테스트하는 효율적인 방법을 ...프로그램 작성 2024년 11월 18일에 게시됨 -
 MySQL 업데이트 쿼리는 기존 값이 동일한 경우 덮어쓰나요?MySQL 업데이트 쿼리: 기존 값 덮어쓰기MySQL에서 테이블을 업데이트할 때 열에 지정하는 새 값이 다음과 같은 시나리오에 직면할 수 있습니다. 현재 가치와 같습니다. 그러한 경우에는 자연스러운 질문이 생깁니다. MySQL이 기존 값을 덮어쓸 것인가, 아니면 업데이...프로그램 작성 2024년 11월 18일에 게시됨
MySQL 업데이트 쿼리는 기존 값이 동일한 경우 덮어쓰나요?MySQL 업데이트 쿼리: 기존 값 덮어쓰기MySQL에서 테이블을 업데이트할 때 열에 지정하는 새 값이 다음과 같은 시나리오에 직면할 수 있습니다. 현재 가치와 같습니다. 그러한 경우에는 자연스러운 질문이 생깁니다. MySQL이 기존 값을 덮어쓸 것인가, 아니면 업데이...프로그램 작성 2024년 11월 18일에 게시됨 -
 `std::atomic`의 저장소가 x86에서 순차적 일관성을 위해 XCHG를 사용하는 이유는 무엇입니까?std::atomic의 매장에서 순차적 일관성을 위해 XCHG를 사용하는 이유x86 및 x86_64 아키텍처에 대한 std::atomic의 맥락에서 순차 일관성을 갖는 저장 작업(std::memory_order_seq_cst)은 순차 릴리스 의미 체계를 달성하기 위한 ...프로그램 작성 2024년 11월 18일에 게시됨
`std::atomic`의 저장소가 x86에서 순차적 일관성을 위해 XCHG를 사용하는 이유는 무엇입니까?std::atomic의 매장에서 순차적 일관성을 위해 XCHG를 사용하는 이유x86 및 x86_64 아키텍처에 대한 std::atomic의 맥락에서 순차 일관성을 갖는 저장 작업(std::memory_order_seq_cst)은 순차 릴리스 의미 체계를 달성하기 위한 ...프로그램 작성 2024년 11월 18일에 게시됨 -
 C++가 함수에서 배열 반환을 직접 지원하지 않는 이유는 무엇입니까?C가 배열 반환 함수를 승인하지 않는 이유C 환경Java와 같은 언어와 달리 C는 배열을 반환하는 함수에 대한 직접적인 지원을 제공하지 않습니다. 배열을 반환할 수 있지만 프로세스가 번거롭습니다. 이는 이 설계 결정의 기본 이유에 대한 의문을 제기합니다.C의 배열 메커...프로그램 작성 2024년 11월 18일에 게시됨
C++가 함수에서 배열 반환을 직접 지원하지 않는 이유는 무엇입니까?C가 배열 반환 함수를 승인하지 않는 이유C 환경Java와 같은 언어와 달리 C는 배열을 반환하는 함수에 대한 직접적인 지원을 제공하지 않습니다. 배열을 반환할 수 있지만 프로세스가 번거롭습니다. 이는 이 설계 결정의 기본 이유에 대한 의문을 제기합니다.C의 배열 메커...프로그램 작성 2024년 11월 18일에 게시됨 -
 좋습니다. 기사 내용에 적합한 제목은 다음과 같습니다. * Qt에서 \"-lGL: 찾을 수 없음\" 오류를 수정하는 방법 * Qt 컴파일 오류: \"-lGL: 찾을 수 없음\" - 해결 방법 * Qt 프로젝트의 \"-lGL: 찾을 수 없음\" 오류 문제 해결 *해결Qt에서 "-lGL: 찾을 수 없음" 오류 해결QtCreator에서 새로 생성된 프로젝트를 컴파일하려고 할 때 일부 사용자에게 다음과 같은 문제가 발생할 수 있습니다. "-lGL: 찾을 수 없음" 오류. 이 오류는 일반적으로 필요한 ...프로그램 작성 2024년 11월 18일에 게시됨
좋습니다. 기사 내용에 적합한 제목은 다음과 같습니다. * Qt에서 \"-lGL: 찾을 수 없음\" 오류를 수정하는 방법 * Qt 컴파일 오류: \"-lGL: 찾을 수 없음\" - 해결 방법 * Qt 프로젝트의 \"-lGL: 찾을 수 없음\" 오류 문제 해결 *해결Qt에서 "-lGL: 찾을 수 없음" 오류 해결QtCreator에서 새로 생성된 프로젝트를 컴파일하려고 할 때 일부 사용자에게 다음과 같은 문제가 발생할 수 있습니다. "-lGL: 찾을 수 없음" 오류. 이 오류는 일반적으로 필요한 ...프로그램 작성 2024년 11월 18일에 게시됨 -
 PHP의 `eval` 기능을 사용해도 안전한가요?평가가 악이 아닌 경우는 언제입니까?PHP의 평가 기능은 종종 권장되지 않지만 PHP 5.3에서의 유용성은 논쟁의 여지가 있습니다. . LSB 및 클로저의 출현에도 불구하고 eval이 여전히 선호되는 몇 가지 사용 사례는 다음과 같습니다.안전한 표현식 평가:Eval은 ...프로그램 작성 2024년 11월 18일에 게시됨
PHP의 `eval` 기능을 사용해도 안전한가요?평가가 악이 아닌 경우는 언제입니까?PHP의 평가 기능은 종종 권장되지 않지만 PHP 5.3에서의 유용성은 논쟁의 여지가 있습니다. . LSB 및 클로저의 출현에도 불구하고 eval이 여전히 선호되는 몇 가지 사용 사례는 다음과 같습니다.안전한 표현식 평가:Eval은 ...프로그램 작성 2024년 11월 18일에 게시됨 -
 macOS의 Django에서 \"부적절하게 구성됨: MySQLdb 모듈 로드 오류\"를 수정하는 방법은 무엇입니까?MySQL이 잘못 구성됨: 상대 경로 문제Django에서 python prepare.py runserver를 실행할 때 다음 오류가 발생할 수 있습니다:ImproperlyConfigured: Error loading MySQLdb module: dlopen(/Libra...프로그램 작성 2024년 11월 18일에 게시됨
macOS의 Django에서 \"부적절하게 구성됨: MySQLdb 모듈 로드 오류\"를 수정하는 방법은 무엇입니까?MySQL이 잘못 구성됨: 상대 경로 문제Django에서 python prepare.py runserver를 실행할 때 다음 오류가 발생할 수 있습니다:ImproperlyConfigured: Error loading MySQLdb module: dlopen(/Libra...프로그램 작성 2024년 11월 18일에 게시됨 -
 Go에서 동적 속성을 사용하여 XML을 역정렬화하는 방법은 무엇입니까?Golang: 동적 속성을 사용하여 XML 역마샬링소개:Go에서 인코딩/xml은 XML 데이터를 처리하는 효율적이고 다양한 방법입니다. 그러나 동적 속성이 있는 XML 요소를 처리할 때 존재하는 속성의 수와 유형을 알 수 없기 때문에 역마샬링이 어려워집니다.질문:XML...프로그램 작성 2024년 11월 18일에 게시됨
Go에서 동적 속성을 사용하여 XML을 역정렬화하는 방법은 무엇입니까?Golang: 동적 속성을 사용하여 XML 역마샬링소개:Go에서 인코딩/xml은 XML 데이터를 처리하는 효율적이고 다양한 방법입니다. 그러나 동적 속성이 있는 XML 요소를 처리할 때 존재하는 속성의 수와 유형을 알 수 없기 때문에 역마샬링이 어려워집니다.질문:XML...프로그램 작성 2024년 11월 18일에 게시됨
중국어 공부
- 1 "걷다"를 중국어로 어떻게 말하나요? 走路 중국어 발음, 走路 중국어 학습
- 2 "비행기를 타다"를 중국어로 어떻게 말하나요? 坐飞机 중국어 발음, 坐飞机 중국어 학습
- 3 "기차를 타다"를 중국어로 어떻게 말하나요? 坐火车 중국어 발음, 坐火车 중국어 학습
- 4 "버스를 타다"를 중국어로 어떻게 말하나요? 坐车 중국어 발음, 坐车 중국어 학습
- 5 운전을 중국어로 어떻게 말하나요? 开车 중국어 발음, 开车 중국어 학습
- 6 수영을 중국어로 뭐라고 하나요? 游泳 중국어 발음, 游泳 중국어 학습
- 7 자전거를 타다 중국어로 뭐라고 하나요? 骑自行车 중국어 발음, 骑自行车 중국어 학습
- 8 중국어로 안녕하세요를 어떻게 말해요? 你好중국어 발음, 你好중국어 학습
- 9 감사합니다를 중국어로 어떻게 말하나요? 谢谢중국어 발음, 谢谢중국어 학습
- 10 How to say goodbye in Chinese? 再见Chinese pronunciation, 再见Chinese learning

























