 Primeira página > Programação > Seleção de recursos com o algoritmo IAMB: um mergulho casual no aprendizado de máquina
Primeira página > Programação > Seleção de recursos com o algoritmo IAMB: um mergulho casual no aprendizado de máquina
Seleção de recursos com o algoritmo IAMB: um mergulho casual no aprendizado de máquina
Então, aqui está a história: recentemente trabalhei em um trabalho escolar do professor Zhuang envolvendo um algoritmo muito legal chamado Incremental Association Markov Blanket (IAMB). Bem, não tenho formação em ciência de dados ou estatística, então este é um território novo para mim, mas adoro aprender algo novo. O objetivo? Use o IAMB para selecionar recursos em um conjunto de dados e ver como isso afeta o desempenho de um modelo de aprendizado de máquina.
Abordaremos os fundamentos do algoritmo IAMB e o aplicaremos ao Pima Indians Diabetes Dataset dos conjuntos de dados de Jason Brownlee. Este conjunto de dados rastreia dados de saúde de mulheres e inclui se elas têm diabetes ou não. Usaremos o IAMB para descobrir quais recursos (como IMC ou níveis de glicose) são mais importantes para prever o diabetes.
O que é o algoritmo IAMB e por que usá-lo?
O algoritmo IAMB é como um amigo que ajuda você a limpar uma lista de suspeitos de um mistério – é um método de seleção de recursos projetado para escolher apenas as variáveis que realmente importam para prever seu alvo. Nesse caso, o objetivo é saber se alguém tem diabetes.
- Fase de avanço: Adicione variáveis que estejam fortemente relacionadas ao alvo.
- Fase inversa: elimine as variáveis que realmente não ajudam, garantindo que apenas as mais cruciais sejam deixadas.
Em termos mais simples, o IAMB nos ajuda a evitar confusão em nosso conjunto de dados, selecionando apenas os recursos mais relevantes. Isso é especialmente útil quando você deseja manter as coisas simples, aumentar o desempenho do modelo e acelerar o tempo de treinamento.
Fonte: Algoritmos para descoberta de manta Markov em grande escala
O que é essa coisa alfa e por que isso importa?
É aqui que entra alfa. Nas estatísticas, alfa (α) é o limite que definimos para decidir o que conta como "estatisticamente significativo". Como parte das instruções dadas pelo professor, usei um alfa de 0,05, o que significa que só quero manter recursos que tenham menos de 5% de chance de serem associados aleatoriamente à variável alvo. Portanto, se o p-valor de um recurso for inferior a 0,05, significa que há uma associação forte e estatisticamente significativa com nosso alvo.
Ao usar esse limite alfa, estamos nos concentrando apenas nas variáveis mais significativas, ignorando aquelas que não passam em nosso teste de “significância”. É como um filtro que mantém os recursos mais relevantes e elimina o ruído.
Colocando a mão na massa: usando IAMB no conjunto de dados de diabetes dos índios Pima
Aqui está a configuração: o conjunto de dados de diabetes dos índios Pima tem recursos de saúde (pressão arterial, idade, níveis de insulina, etc.) e nossa meta, Resultado (se alguém tem diabetes).
Primeiro, carregamos os dados e verificamos:
import pandas as pd # Load and preview the dataset url = 'https://raw.githubusercontent.com/jbrownlee/Datasets/master/pima-indians-diabetes.data.csv' column_names = ['Pregnancies', 'Glucose', 'BloodPressure', 'SkinThickness', 'Insulin', 'BMI', 'DiabetesPedigreeFunction', 'Age', 'Outcome'] data = pd.read_csv(url, names=column_names) print(data.head())
Implementando IAMB com Alpha = 0,05
Aqui está nossa versão atualizada do algoritmo IAMB. Estamos usando valores de p para decidir quais recursos manter, portanto, apenas aqueles com valores de p menores que nosso alfa (0,05) são selecionados.
import pingouin as pg
def iamb(target, data, alpha=0.05):
markov_blanket = set()
# Forward Phase: Add features with a p-value alpha
for feature in list(markov_blanket):
reduced_mb = markov_blanket - {feature}
result = pg.partial_corr(data=data, x=feature, y=target, covar=reduced_mb)
p_value = result.at[0, 'p-val']
if p_value > alpha:
markov_blanket.remove(feature)
return list(markov_blanket)
# Apply the updated IAMB function on the Pima dataset
selected_features = iamb('Outcome', data, alpha=0.05)
print("Selected Features:", selected_features)
Quando executei isso, obtive uma lista refinada de recursos que o IAMB considerou estarem mais intimamente relacionados aos resultados do diabetes. Esta lista ajuda a restringir as variáveis que precisamos para construir nosso modelo.
Selected Features: ['BMI', 'DiabetesPedigreeFunction', 'Pregnancies', 'Glucose']
Testando o impacto dos recursos selecionados pelo IAMB no desempenho do modelo
Assim que tivermos nossos recursos selecionados, o teste real compara o desempenho do modelo com todos os recursos versus recursos selecionados pelo IAMB. Para isso, optei por um modelo simples Gaussian Naive Bayes porque é direto e funciona bem com probabilidades (o que está de acordo com toda a vibração bayesiana).
Aqui está o código para treinar e testar o modelo:
from sklearn.model_selection import train_test_split
from sklearn.naive_bayes import GaussianNB
from sklearn.metrics import accuracy_score, f1_score, roc_auc_score
# Split data
X = data.drop('Outcome', axis=1)
y = data['Outcome']
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.3, random_state=42)
# Model with All Features
model_all = GaussianNB()
model_all.fit(X_train, y_train)
y_pred_all = model_all.predict(X_test)
# Model with IAMB-Selected Features
X_train_selected = X_train[selected_features]
X_test_selected = X_test[selected_features]
model_iamb = GaussianNB()
model_iamb.fit(X_train_selected, y_train)
y_pred_iamb = model_iamb.predict(X_test_selected)
# Evaluate models
results = {
'Model': ['All Features', 'IAMB-Selected Features'],
'Accuracy': [accuracy_score(y_test, y_pred_all), accuracy_score(y_test, y_pred_iamb)],
'F1 Score': [f1_score(y_test, y_pred_all, average='weighted'), f1_score(y_test, y_pred_iamb, average='weighted')],
'AUC-ROC': [roc_auc_score(y_test, y_pred_all), roc_auc_score(y_test, y_pred_iamb)]
}
results_df = pd.DataFrame(results)
display(results_df)
Resultados
Esta é a comparação:
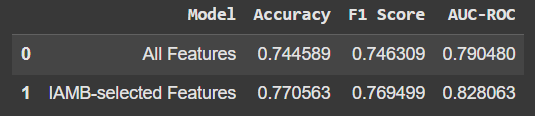
Usar apenas os recursos selecionados pelo IAMB proporcionou um ligeiro aumento na precisão e em outras métricas. Não é um grande salto, mas o fato de estarmos obtendo melhor desempenho com menos recursos é promissor. Além disso, significa que nosso modelo não depende de “ruído” ou dados irrelevantes.
Principais conclusões
- IAMB é ótimo para seleção de recursos: ajuda a limpar nosso conjunto de dados, concentrando-se apenas no que realmente importa para prever nosso alvo.
- Menos geralmente é mais: Às vezes, menos recursos nos dão melhores resultados, como vimos aqui com um pequeno aumento na precisão do modelo.
- Aprender e experimentar é a parte divertida: Mesmo sem uma profunda experiência em ciência de dados, mergulhar em projetos como este abre novas maneiras de entender os dados e o aprendizado de máquina.
Espero que isso forneça uma introdução amigável ao IAMB! Se você estiver curioso, experimente: é uma ferramenta útil na caixa de ferramentas de aprendizado de máquina e você poderá ver algumas melhorias interessantes em seus próprios projetos.
Fonte: Algoritmos para descoberta de manta Markov em grande escala
-
 Explorando a força da senha e a validação numérica em Perl and GoNeste artigo, abordarei dois desafios do Perl Weekly Challenge #287: fortalecer senhas fracas e validar números. Fornecerei soluções para ambas as tar...Programação Publicado em 2024-11-18
Explorando a força da senha e a validação numérica em Perl and GoNeste artigo, abordarei dois desafios do Perl Weekly Challenge #287: fortalecer senhas fracas e validar números. Fornecerei soluções para ambas as tar...Programação Publicado em 2024-11-18 -
 Além das instruções `if`: onde mais um tipo com uma conversão `bool` explícita pode ser usado sem conversão?Conversão contextual para bool permitida sem conversãoSua classe define uma conversão explícita para bool, permitindo que você use sua instância '...Programação Publicado em 2024-11-18
Além das instruções `if`: onde mais um tipo com uma conversão `bool` explícita pode ser usado sem conversão?Conversão contextual para bool permitida sem conversãoSua classe define uma conversão explícita para bool, permitindo que você use sua instância '...Programação Publicado em 2024-11-18 -
 Como escrever APIs como um profissional usando GopherLightDocumentos GopherLIght Olá pessoal, primeiro gostaria de agradecer por escolherem usar nosso projeto. Mesmo sendo pequeno, fizemos isso com ...Programação Publicado em 2024-11-18
Como escrever APIs como um profissional usando GopherLightDocumentos GopherLIght Olá pessoal, primeiro gostaria de agradecer por escolherem usar nosso projeto. Mesmo sendo pequeno, fizemos isso com ...Programação Publicado em 2024-11-18 -
 O que aconteceu com o deslocamento de coluna no Bootstrap 4 Beta?Bootstrap 4 Beta: A remoção e restauração do deslocamento de colunaBootstrap 4, em sua versão Beta 1, introduziu mudanças significativas na forma como...Programação Publicado em 2024-11-18
O que aconteceu com o deslocamento de coluna no Bootstrap 4 Beta?Bootstrap 4 Beta: A remoção e restauração do deslocamento de colunaBootstrap 4, em sua versão Beta 1, introduziu mudanças significativas na forma como...Programação Publicado em 2024-11-18 -
 Como corrigir “Configuração incorreta: Erro ao carregar o módulo MySQLdb” no Django no macOS?MySQL configurado incorretamente: o problema com caminhos relativosAo executar python manage.py runserver no Django, você pode encontrar o seguinte er...Programação Publicado em 2024-11-18
Como corrigir “Configuração incorreta: Erro ao carregar o módulo MySQLdb” no Django no macOS?MySQL configurado incorretamente: o problema com caminhos relativosAo executar python manage.py runserver no Django, você pode encontrar o seguinte er...Programação Publicado em 2024-11-18 -
 Como combinar dois arrays associativos em PHP preservando IDs exclusivos e manipulando nomes duplicados?Combinando matrizes associativas em PHPEm PHP, combinar duas matrizes associativas em uma única matriz é uma tarefa comum. Considere a seguinte solici...Programação Publicado em 2024-11-18
Como combinar dois arrays associativos em PHP preservando IDs exclusivos e manipulando nomes duplicados?Combinando matrizes associativas em PHPEm PHP, combinar duas matrizes associativas em uma única matriz é uma tarefa comum. Considere a seguinte solici...Programação Publicado em 2024-11-18 -
 Como posso encontrar usuários com aniversários de hoje usando MySQL?Como identificar usuários com aniversários de hoje usando MySQLDeterminar se hoje é o aniversário de um usuário usando MySQL envolve encontrar todas a...Programação Publicado em 2024-11-18
Como posso encontrar usuários com aniversários de hoje usando MySQL?Como identificar usuários com aniversários de hoje usando MySQLDeterminar se hoje é o aniversário de um usuário usando MySQL envolve encontrar todas a...Programação Publicado em 2024-11-18 -
 Como posso usar variáveis CSS sem unidade com unidades diferentes?Como usar variáveis CSS sem unidade com flexibilidadeAs variáveis CSS sem unidade fornecem a capacidade de armazenar valores numéricos que podem s...Programação Publicado em 2024-11-18
Como posso usar variáveis CSS sem unidade com unidades diferentes?Como usar variáveis CSS sem unidade com flexibilidadeAs variáveis CSS sem unidade fornecem a capacidade de armazenar valores numéricos que podem s...Programação Publicado em 2024-11-18 -
 Executando uma função quando um bloco #await é resolvido em Svelte(Kit)Pular para o conteúdo: Sobre o bloco #await em svelte Execute (acione) uma função quando o bloco #await for resolvido ou rejeitado Corrigir texto inde...Programação Publicado em 2024-11-18
Executando uma função quando um bloco #await é resolvido em Svelte(Kit)Pular para o conteúdo: Sobre o bloco #await em svelte Execute (acione) uma função quando o bloco #await for resolvido ou rejeitado Corrigir texto inde...Programação Publicado em 2024-11-18 -
 Você pode ter várias classes em um único arquivo Java?Várias classes em um arquivo JavaEm Java, é possível ter várias classes em um único arquivo .java. No entanto, só pode haver uma classe pública de nív...Programação Publicado em 2024-11-18
Você pode ter várias classes em um único arquivo Java?Várias classes em um arquivo JavaEm Java, é possível ter várias classes em um único arquivo .java. No entanto, só pode haver uma classe pública de nív...Programação Publicado em 2024-11-18 -
 Como testar conexões de banco de dados PDO e lidar com erros de maneira eficaz?Testando conexões de banco de dados PDOAo desenvolver instalações de banco de dados, é crucial garantir a validade das conexões de banco de dados. Ist...Programação Publicado em 2024-11-18
Como testar conexões de banco de dados PDO e lidar com erros de maneira eficaz?Testando conexões de banco de dados PDOAo desenvolver instalações de banco de dados, é crucial garantir a validade das conexões de banco de dados. Ist...Programação Publicado em 2024-11-18 -
 As consultas de atualização do MySQL substituem os valores existentes quando eles são iguais?Consultas de atualização do MySQL: substituindo valores existentesNo MySQL, ao atualizar uma tabela, é possível encontrar um cenário em que o novo val...Programação Publicado em 2024-11-18
As consultas de atualização do MySQL substituem os valores existentes quando eles são iguais?Consultas de atualização do MySQL: substituindo valores existentesNo MySQL, ao atualizar uma tabela, é possível encontrar um cenário em que o novo val...Programação Publicado em 2024-11-18 -
 Por que o armazenamento `std::atomic` usa XCHG para consistência sequencial em x86?Por que a loja std::atomic emprega XCHG para consistência sequencialNo contexto de std::atomic para arquiteturas x86 e x86_64, um operação de armazena...Programação Publicado em 2024-11-18
Por que o armazenamento `std::atomic` usa XCHG para consistência sequencial em x86?Por que a loja std::atomic emprega XCHG para consistência sequencialNo contexto de std::atomic para arquiteturas x86 e x86_64, um operação de armazena...Programação Publicado em 2024-11-18 -
 VariedadeMétodos são fns que podem ser chamados em objetos Arrays são objetos, portanto também possuem métodos em JS. slice(begin): extrai parte do arr...Programação Publicado em 2024-11-18
VariedadeMétodos são fns que podem ser chamados em objetos Arrays são objetos, portanto também possuem métodos em JS. slice(begin): extrai parte do arr...Programação Publicado em 2024-11-18 -
 Por que o C++ não suporta diretamente o retorno de matrizes de funções?Por que C desaprova funções de retorno de arrayO cenário CEm contraste com linguagens como Java, C não não oferece suporte direto para funções que ret...Programação Publicado em 2024-11-18
Por que o C++ não suporta diretamente o retorno de matrizes de funções?Por que C desaprova funções de retorno de arrayO cenário CEm contraste com linguagens como Java, C não não oferece suporte direto para funções que ret...Programação Publicado em 2024-11-18
Estude chinês
- 1 Como se diz “andar” em chinês? 走路 Pronúncia chinesa, 走路 aprendizagem chinesa
- 2 Como se diz “pegar um avião” em chinês? 坐飞机 Pronúncia chinesa, 坐飞机 aprendizagem chinesa
- 3 Como se diz “pegar um trem” em chinês? 坐火车 Pronúncia chinesa, 坐火车 aprendizagem chinesa
- 4 Como se diz “pegar um ônibus” em chinês? 坐车 Pronúncia chinesa, 坐车 aprendizagem chinesa
- 5 Como se diz dirigir em chinês? 开车 Pronúncia chinesa, 开车 aprendizagem chinesa
- 6 Como se diz nadar em chinês? 游泳 Pronúncia chinesa, 游泳 aprendizagem chinesa
- 7 Como se diz andar de bicicleta em chinês? 骑自行车 Pronúncia chinesa, 骑自行车 aprendizagem chinesa
- 8 Como você diz olá em chinês? 你好Pronúncia chinesa, 你好Aprendizagem chinesa
- 9 Como você agradece em chinês? 谢谢Pronúncia chinesa, 谢谢Aprendizagem chinesa
- 10 How to say goodbye in Chinese? 再见Chinese pronunciation, 再见Chinese learning
























