 मुखपृष्ठ > प्रोग्रामिंग > छवि विभाजन में महारत हासिल करना: डिजिटल युग में पारंपरिक तकनीकें अभी भी कैसे चमक रही हैं
मुखपृष्ठ > प्रोग्रामिंग > छवि विभाजन में महारत हासिल करना: डिजिटल युग में पारंपरिक तकनीकें अभी भी कैसे चमक रही हैं
छवि विभाजन में महारत हासिल करना: डिजिटल युग में पारंपरिक तकनीकें अभी भी कैसे चमक रही हैं
परिचय
छवि विभाजन, कंप्यूटर दृष्टि में सबसे बुनियादी प्रक्रियाओं में से एक, एक प्रणाली को एक छवि के भीतर विभिन्न क्षेत्रों को विघटित और विश्लेषण करने की अनुमति देता है। चाहे आप वस्तु पहचान, चिकित्सा इमेजिंग, या स्वायत्त ड्राइविंग से निपट रहे हों, विभाजन ही छवियों को सार्थक भागों में तोड़ता है।
हालांकि इस कार्य में गहन शिक्षण मॉडल तेजी से लोकप्रिय हो रहे हैं, डिजिटल इमेज प्रोसेसिंग में पारंपरिक तकनीक अभी भी शक्तिशाली और व्यावहारिक हैं। इस पोस्ट में जिन दृष्टिकोणों की समीक्षा की जा रही है उनमें सेल छवियों के विश्लेषण के लिए एक अच्छी तरह से मान्यता प्राप्त डेटासेट, MIVIA HEp-2 इमेज डेटासेट को लागू करके थ्रेशोल्डिंग, एज डिटेक्शन, क्षेत्र-आधारित और क्लस्टरिंग शामिल है।
MIVIA HEp-2 छवि डेटासेट
MIVIA HEp-2 इमेज डेटासेट कोशिकाओं के चित्रों का एक सेट है जिसका उपयोग HEp-2 कोशिकाओं के माध्यम से एंटीन्यूक्लियर एंटीबॉडी (ANA) के पैटर्न का विश्लेषण करने के लिए किया जाता है। इसमें प्रतिदीप्ति माइक्रोस्कोपी के माध्यम से ली गई 2डी तस्वीरें शामिल हैं। यह इसे विभाजन कार्यों के लिए बहुत उपयुक्त बनाता है, सबसे महत्वपूर्ण रूप से चिकित्सा छवि विश्लेषण से संबंधित कार्यों के लिए, जहां सेलुलर क्षेत्र का पता लगाना सबसे महत्वपूर्ण है।
अब, आइए इन छवियों को संसाधित करने के लिए उपयोग की जाने वाली विभाजन तकनीकों पर आगे बढ़ें, एफ1 स्कोर के आधार पर उनके प्रदर्शन की तुलना करें।
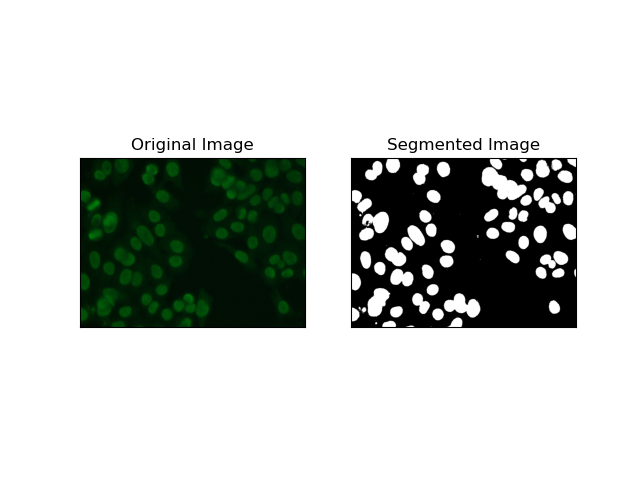
1. दहलीज विभाजन
थ्रेसहोल्डिंग वह प्रक्रिया है जिसके तहत ग्रेस्केल छवियां पिक्सेल तीव्रता के आधार पर बाइनरी छवियों में परिवर्तित हो जाती हैं। MIVIA HEp-2 डेटासेट में, यह प्रक्रिया पृष्ठभूमि से सेल निष्कर्षण में उपयोगी है। यह अपेक्षाकृत बड़े स्तर पर सरल और प्रभावी है, विशेष रूप से ओत्सु की विधि के साथ, क्योंकि यह इष्टतम सीमा की स्व-गणना करता है।
ओत्सु की विधि एक स्वचालित थ्रेशोल्डिंग विधि है, जहां यह न्यूनतम इंट्रा-क्लास विचरण प्राप्त करने के लिए सर्वोत्तम थ्रेशोल्ड मान खोजने की कोशिश करती है, जिससे दो वर्गों को अलग किया जाता है: अग्रभूमि (कोशिकाएं) और पृष्ठभूमि। विधि छवि हिस्टोग्राम की जांच करती है और सही सीमा की गणना करती है, जहां प्रत्येक वर्ग में पिक्सेल तीव्रता भिन्नता का योग कम से कम किया जाता है।
# Thresholding Segmentation
def thresholding(img):
# Convert image to grayscale
gray = cv.cvtColor(img, cv.COLOR_BGR2GRAY)
# Apply Otsu's thresholding
_, thresh = cv.threshold(gray, 0, 255, cv.THRESH_BINARY cv.THRESH_OTSU)
return thresh
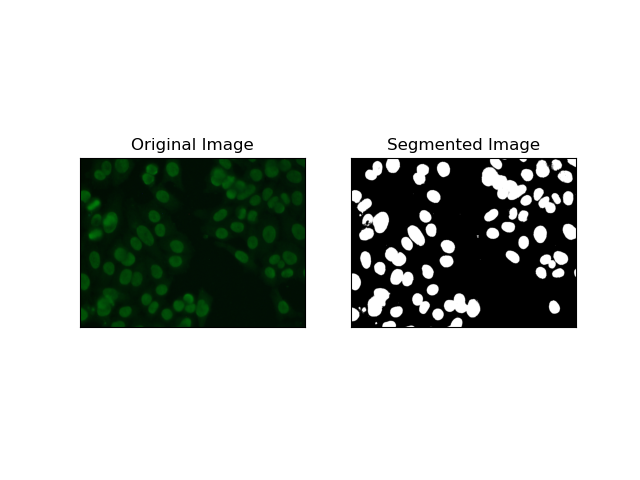
2. एज डिटेक्शन सेगमेंटेशन
किनारे का पता लगाना वस्तुओं या क्षेत्रों की सीमाओं की पहचान करने से संबंधित है, जैसे कि MIVIA HEp-2 डेटासेट में सेल किनारे। अचानक तीव्रता में परिवर्तन का पता लगाने के लिए कई उपलब्ध तरीकों में से, कैनी एज डिटेक्टर सेलुलर सीमाओं का पता लगाने के लिए सबसे अच्छा और इसलिए सबसे उपयुक्त तरीका है।
कैनी एज डिटेक्टर एक मल्टी-स्टेज एल्गोरिदम है जो तीव्रता के मजबूत ग्रेडिएंट वाले क्षेत्रों का पता लगाकर किनारों का पता लगा सकता है। इस प्रक्रिया में गॉसियन फिल्टर के साथ स्मूथिंग, तीव्रता ग्रेडिएंट्स की गणना, नकली प्रतिक्रियाओं को खत्म करने के लिए गैर-अधिकतम दमन का अनुप्रयोग और केवल मुख्य किनारों को बनाए रखने के लिए अंतिम डबल थ्रेशोल्डिंग ऑपरेशन शामिल है।
# Edge Detection Segmentation
def edge_detection(img):
# Convert image to grayscale
gray = cv.cvtColor(img, cv.COLOR_BGR2GRAY)
# Apply Gaussian blur
gray = cv.GaussianBlur(gray, (3, 3), 0)
# Calculate lower and upper thresholds for Canny edge detection
sigma = 0.33
v = np.median(gray)
lower = int(max(0, (1.0 - sigma) * v))
upper = int(min(255, (1.0 sigma) * v))
# Apply Canny edge detection
edges = cv.Canny(gray, lower, upper)
# Dilate the edges to fill gaps
kernel = np.ones((5, 5), np.uint8)
dilated_edges = cv.dilate(edges, kernel, iterations=2)
# Clean the edges using morphological opening
cleaned_edges = cv.morphologyEx(dilated_edges, cv.MORPH_OPEN, kernel, iterations=1)
# Find connected components and filter out small components
num_labels, labels, stats, _ = cv.connectedComponentsWithStats(
cleaned_edges, connectivity=8
)
min_size = 500
filtered_mask = np.zeros_like(cleaned_edges)
for i in range(1, num_labels):
if stats[i, cv.CC_STAT_AREA] >= min_size:
filtered_mask[labels == i] = 255
# Find contours of the filtered mask
contours, _ = cv.findContours(
filtered_mask, cv.RETR_EXTERNAL, cv.CHAIN_APPROX_SIMPLE
)
# Create a filled mask using the contours
filled_mask = np.zeros_like(gray)
cv.drawContours(filled_mask, contours, -1, (255), thickness=cv.FILLED)
# Perform morphological closing to fill holes
final_filled_image = cv.morphologyEx(
filled_mask, cv.MORPH_CLOSE, kernel, iterations=2
)
# Dilate the final filled image to smooth the edges
final_filled_image = cv.dilate(final_filled_image, kernel, iterations=1)
return final_filled_image
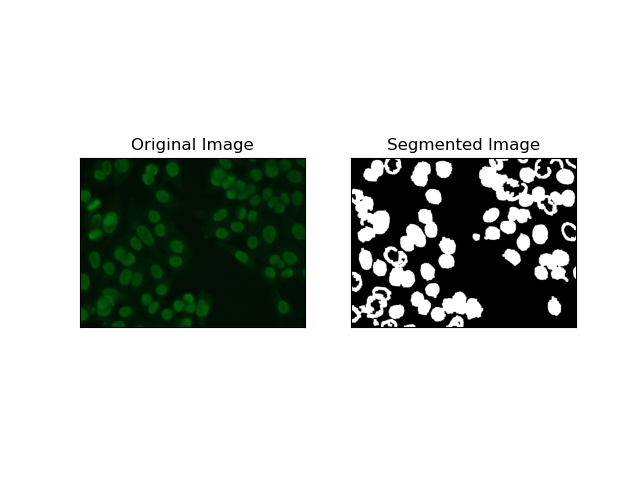
3. क्षेत्र-आधारित विभाजन
क्षेत्र-आधारित विभाजन समान पिक्सेल को क्षेत्रों में एक साथ समूहित करता है, जो तीव्रता या रंग जैसे कुछ मानदंडों पर निर्भर करता है। वाटरशेड विभाजन तकनीक का उपयोग एचईपी-2 सेल छवियों को विभाजित करने में मदद के लिए किया जा सकता है ताकि उन क्षेत्रों का पता लगाया जा सके जो कोशिकाओं का प्रतिनिधित्व करते हैं; यह पिक्सेल तीव्रता को एक स्थलाकृतिक सतह के रूप में मानता है और विशिष्ट क्षेत्रों की रूपरेखा तैयार करता है।
वाटरशेड विभाजन पिक्सेल की तीव्रता को स्थलाकृतिक सतह के रूप में मानता है। एल्गोरिदम "बेसिन" की पहचान करता है जिसमें यह स्थानीय मिनीमा की पहचान करता है और फिर अलग-अलग क्षेत्रों को बड़ा करने के लिए धीरे-धीरे इन बेसिनों में बाढ़ लाता है। यह तकनीक तब काफी उपयोगी होती है जब कोई छूने वाली वस्तुओं को अलग करना चाहता है, जैसे कि सूक्ष्म छवियों के भीतर कोशिकाओं के मामले में, लेकिन यह शोर के प्रति संवेदनशील हो सकती है। प्रक्रिया को मार्करों द्वारा निर्देशित किया जा सकता है और अति-विभाजन को अक्सर कम किया जा सकता है।
# Region-Based Segmentation
def region_based(img):
# Convert image to grayscale
gray = cv.cvtColor(img, cv.COLOR_BGR2GRAY)
# Apply Otsu's thresholding
_, thresh = cv.threshold(gray, 0, 255, cv.THRESH_BINARY_INV cv.THRESH_OTSU)
# Apply morphological opening to remove noise
kernel = np.ones((3, 3), np.uint8)
opening = cv.morphologyEx(thresh, cv.MORPH_OPEN, kernel, iterations=2)
# Dilate the opening to get the background
sure_bg = cv.dilate(opening, kernel, iterations=3)
# Calculate the distance transform
dist_transform = cv.distanceTransform(opening, cv.DIST_L2, 5)
# Threshold the distance transform to get the foreground
_, sure_fg = cv.threshold(dist_transform, 0.2 * dist_transform.max(), 255, 0)
sure_fg = np.uint8(sure_fg)
# Find the unknown region
unknown = cv.subtract(sure_bg, sure_fg)
# Label the markers for watershed algorithm
_, markers = cv.connectedComponents(sure_fg)
markers = markers 1
markers[unknown == 255] = 0
# Apply watershed algorithm
markers = cv.watershed(img, markers)
# Create a mask for the segmented region
mask = np.zeros_like(gray, dtype=np.uint8)
mask[markers == 1] = 255
return mask
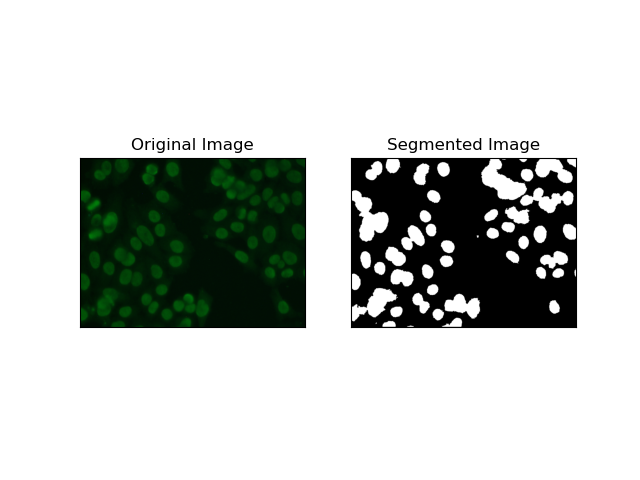
4. क्लस्टरिंग-आधारित विभाजन
क्लस्टरिंग तकनीकें जैसे कि K-Means पिक्सेल को समान समूहों में समूहित करती हैं, जो बहु-रंगीन या जटिल वातावरण में कोशिकाओं को विभाजित करने की इच्छा होने पर ठीक काम करती है, जैसा कि HEp-2 सेल छवियों में देखा जाता है। मौलिक रूप से, यह विभिन्न वर्गों का प्रतिनिधित्व कर सकता है, जैसे सेलुलर क्षेत्र बनाम पृष्ठभूमि।
K-means रंग या तीव्रता की पिक्सेल समानता के आधार पर छवियों को क्लस्टर करने के लिए एक अप्रशिक्षित शिक्षण एल्गोरिदम है। एल्गोरिथ्म बेतरतीब ढंग से K सेंट्रोइड का चयन करता है, प्रत्येक पिक्सेल को निकटतम सेंट्रोइड को निर्दिष्ट करता है, और सेंट्रोइड को तब तक अद्यतन करता है जब तक कि यह अभिसरण न हो जाए। यह ऐसी छवि को खंडित करने में विशेष रूप से प्रभावी है जिसमें रुचि के कई क्षेत्र होते हैं जो एक दूसरे से बहुत भिन्न होते हैं।
# Clustering Segmentation
def clustering(img):
# Convert image to grayscale
gray = cv.cvtColor(img, cv.COLOR_BGR2GRAY)
# Reshape the image
Z = gray.reshape((-1, 3))
Z = np.float32(Z)
# Define the criteria for k-means clustering
criteria = (cv.TERM_CRITERIA_EPS cv.TERM_CRITERIA_MAX_ITER, 10, 1.0)
# Set the number of clusters
K = 2
# Perform k-means clustering
_, label, center = cv.kmeans(Z, K, None, criteria, 10, cv.KMEANS_RANDOM_CENTERS)
# Convert the center values to uint8
center = np.uint8(center)
# Reshape the result
res = center[label.flatten()]
res = res.reshape((gray.shape))
# Apply thresholding to the result
_, res = cv.threshold(res, 0, 255, cv.THRESH_BINARY cv.THRESH_OTSU)
return res
F1 स्कोर का उपयोग करके तकनीकों का मूल्यांकन करना
F1 स्कोर एक माप है जो अनुमानित विभाजन छवि की जमीनी सच्चाई छवि के साथ तुलना करने के लिए सटीकता और रिकॉल को एक साथ जोड़ता है। यह सटीकता और रिकॉल का हार्मोनिक माध्यम है, जो उच्च डेटा असंतुलन के मामलों में उपयोगी है, जैसे कि मेडिकल इमेजिंग डेटासेट में।
हमने जमीनी सच्चाई और खंडित छवि दोनों को समतल करके और भारित एफ1 स्कोर की गणना करके प्रत्येक विभाजन विधि के लिए एफ1 स्कोर की गणना की।
def calculate_f1_score(ground_image, segmented_image):
ground_image = ground_image.flatten()
segmented_image = segmented_image.flatten()
return f1_score(ground_image, segmented_image, average="weighted")
फिर हमने एक साधारण बार चार्ट का उपयोग करके विभिन्न तरीकों के एफ1 स्कोर की कल्पना की:
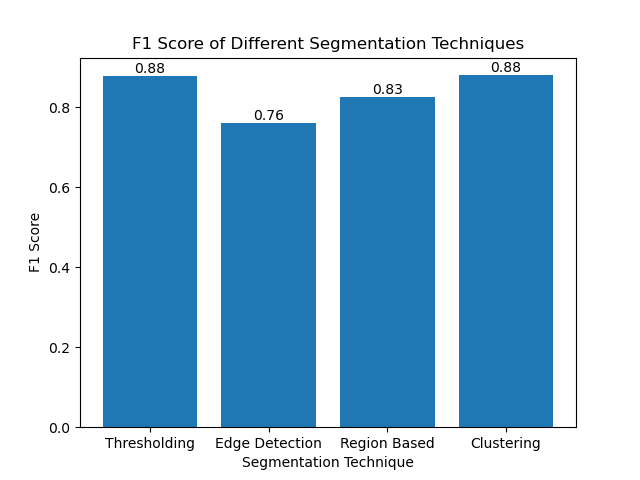
निष्कर्ष
हालांकि छवि विभाजन के लिए कई हालिया दृष्टिकोण उभर रहे हैं, पारंपरिक विभाजन तकनीक जैसे थ्रेशोल्डिंग, एज डिटेक्शन, क्षेत्र-आधारित तरीके और क्लस्टरिंग MIVIA HEp-2 छवि डेटासेट जैसे डेटासेट पर लागू होने पर बहुत उपयोगी हो सकते हैं।
प्रत्येक विधि की अपनी ताकत होती है:
- थ्रेसहोल्डिंग सरल बाइनरी सेगमेंटेशन के लिए अच्छा है।
- एज डिटेक्शन सीमाओं का पता लगाने के लिए एक आदर्श तकनीक है।
- क्षेत्र-आधारित विभाजन जुड़े हुए घटकों को उनके पड़ोसियों से अलग करने में बहुत उपयोगी है।
- क्लस्टरिंग विधियां बहु-क्षेत्रीय विभाजन कार्यों के लिए उपयुक्त हैं।
एफ1 स्कोर का उपयोग करके इन तरीकों का मूल्यांकन करके, हम इनमें से प्रत्येक मॉडल के ट्रेड-ऑफ को समझते हैं। हो सकता है कि ये विधियाँ उतनी परिष्कृत न हों जितनी कि गहन शिक्षण के नवीनतम मॉडलों में विकसित की गई हैं, लेकिन वे अनुप्रयोगों की एक विस्तृत श्रृंखला में अभी भी तेज़, व्याख्या योग्य और सेवा योग्य हैं।
पढ़ने के लिए धन्यवाद! मुझे आशा है कि पारंपरिक छवि विभाजन तकनीकों की यह खोज आपके अगले प्रोजेक्ट को प्रेरित करेगी। नीचे टिप्पणी में अपने विचार और अनुभव बेझिझक साझा करें!
-
 मुझे अपनी सिल्वरलाइट LINQ क्वेरी में "क्वेरी पैटर्न का कार्यान्वयन" त्रुटि क्यों नहीं मिल रही है?] यह त्रुटि आम तौर पर तब होती है जब या तो Linq नेमस्पेस को छोड़ दिया जाता है या queried प्रकार में ienumerable कार्यान्वयन का अभाव होता है। इस विशिष्...प्रोग्रामिंग 2025-04-06 पर पोस्ट किया गया
मुझे अपनी सिल्वरलाइट LINQ क्वेरी में "क्वेरी पैटर्न का कार्यान्वयन" त्रुटि क्यों नहीं मिल रही है?] यह त्रुटि आम तौर पर तब होती है जब या तो Linq नेमस्पेस को छोड़ दिया जाता है या queried प्रकार में ienumerable कार्यान्वयन का अभाव होता है। इस विशिष्...प्रोग्रामिंग 2025-04-06 पर पोस्ट किया गया -
 मैं PHP के फाइलसिस्टम फ़ंक्शंस में UTF-8 फ़ाइल नाम कैसे संभाल सकता हूं?असंगतता। mkdir ($ dir_name); मूल UTF-8 फ़ाइल नाम को पुनः प्राप्त करने के लिए, urldecode का उपयोग करें। केवल) विंडोज पर, आप UTF-8 फ़ाइल नाम ...प्रोग्रामिंग 2025-04-06 पर पोस्ट किया गया
मैं PHP के फाइलसिस्टम फ़ंक्शंस में UTF-8 फ़ाइल नाम कैसे संभाल सकता हूं?असंगतता। mkdir ($ dir_name); मूल UTF-8 फ़ाइल नाम को पुनः प्राप्त करने के लिए, urldecode का उपयोग करें। केवल) विंडोज पर, आप UTF-8 फ़ाइल नाम ...प्रोग्रामिंग 2025-04-06 पर पोस्ट किया गया -
 पीडीओ मापदंडों के साथ क्वेरी की तरह सही तरीके से उपयोग कैसे करें?$ params = सरणी ($ var1, $ var2); $ stmt = $ हैंडल-> तैयार करें ($ क्वेरी); $ stmt-> निष्पादित ($ params); त्रुटि % संकेतों के गलत समावेश में निहित ह...प्रोग्रामिंग 2025-04-06 पर पोस्ट किया गया
पीडीओ मापदंडों के साथ क्वेरी की तरह सही तरीके से उपयोग कैसे करें?$ params = सरणी ($ var1, $ var2); $ stmt = $ हैंडल-> तैयार करें ($ क्वेरी); $ stmt-> निष्पादित ($ params); त्रुटि % संकेतों के गलत समावेश में निहित ह...प्रोग्रामिंग 2025-04-06 पर पोस्ट किया गया -
 Ubuntu/linux पर mysql-python स्थापित करते समय \ "mysql_config को कैसे नहीं मिला \" त्रुटि नहीं मिली?] यह त्रुटि एक लापता MySQL विकास पुस्तकालय के कारण उत्पन्न होती है। निम्नलिखित कमांड का उपयोग करके पायथन-mysqldb स्थापित करें: sudo apt-get python-...प्रोग्रामिंग 2025-04-06 पर पोस्ट किया गया
Ubuntu/linux पर mysql-python स्थापित करते समय \ "mysql_config को कैसे नहीं मिला \" त्रुटि नहीं मिली?] यह त्रुटि एक लापता MySQL विकास पुस्तकालय के कारण उत्पन्न होती है। निम्नलिखित कमांड का उपयोग करके पायथन-mysqldb स्थापित करें: sudo apt-get python-...प्रोग्रामिंग 2025-04-06 पर पोस्ट किया गया -
 गो में SQL प्रश्नों का निर्माण करते समय मैं सुरक्षित रूप से पाठ और मूल्यों को कैसे सहमत कर सकता हूं?] दृष्टिकोण जाने में मान्य नहीं है, और मापदंडों को कास्ट करने का प्रयास करने के लिए स्ट्रिंग्स के परिणामस्वरूप बेमेल त्रुटियां होती हैं। यह आपको रनटाइ...प्रोग्रामिंग 2025-04-06 पर पोस्ट किया गया
गो में SQL प्रश्नों का निर्माण करते समय मैं सुरक्षित रूप से पाठ और मूल्यों को कैसे सहमत कर सकता हूं?] दृष्टिकोण जाने में मान्य नहीं है, और मापदंडों को कास्ट करने का प्रयास करने के लिए स्ट्रिंग्स के परिणामस्वरूप बेमेल त्रुटियां होती हैं। यह आपको रनटाइ...प्रोग्रामिंग 2025-04-06 पर पोस्ट किया गया -
 जावा के पूर्ण-स्क्रीन अनन्य मोड में उपयोगकर्ता इनपुट को कैसे संभालें?java में पूर्ण स्क्रीन अनन्य मोड में उपयोगकर्ता इनपुट को संभालना, जब पूर्ण स्क्रीन अनन्य मोड में एक जावा एप्लिकेशन चलाना अपेक्षित नहीं हो ...प्रोग्रामिंग 2025-04-06 पर पोस्ट किया गया
जावा के पूर्ण-स्क्रीन अनन्य मोड में उपयोगकर्ता इनपुट को कैसे संभालें?java में पूर्ण स्क्रीन अनन्य मोड में उपयोगकर्ता इनपुट को संभालना, जब पूर्ण स्क्रीन अनन्य मोड में एक जावा एप्लिकेशन चलाना अपेक्षित नहीं हो ...प्रोग्रामिंग 2025-04-06 पर पोस्ट किया गया -
 Google API से नवीनतम JQuery लाइब्रेरी कैसे पुनः प्राप्त करें?] नवीनतम संस्करण को पुनर्प्राप्त करने के लिए, पहले एक विशिष्ट संस्करण संख्या का उपयोग करने का एक विकल्प था, जो निम्न सिंटैक्स का उपयोग करना था: htt...प्रोग्रामिंग 2025-04-06 पर पोस्ट किया गया
Google API से नवीनतम JQuery लाइब्रेरी कैसे पुनः प्राप्त करें?] नवीनतम संस्करण को पुनर्प्राप्त करने के लिए, पहले एक विशिष्ट संस्करण संख्या का उपयोग करने का एक विकल्प था, जो निम्न सिंटैक्स का उपयोग करना था: htt...प्रोग्रामिंग 2025-04-06 पर पोस्ट किया गया -
 मैं सेल एडिटिंग के बाद कस्टम जेटेबल सेल रेंडरिंग कैसे बनाए रख सकता हूं?हालाँकि, यह सुनिश्चित करना महत्वपूर्ण है कि वांछित स्वरूपण को संपादन संचालन के बाद भी संरक्षित किया गया है। इस तरह के परिदृश्यों में, सेल रेंडरर का ड...प्रोग्रामिंग 2025-04-06 पर पोस्ट किया गया
मैं सेल एडिटिंग के बाद कस्टम जेटेबल सेल रेंडरिंग कैसे बनाए रख सकता हूं?हालाँकि, यह सुनिश्चित करना महत्वपूर्ण है कि वांछित स्वरूपण को संपादन संचालन के बाद भी संरक्षित किया गया है। इस तरह के परिदृश्यों में, सेल रेंडरर का ड...प्रोग्रामिंग 2025-04-06 पर पोस्ट किया गया -
 मैं फॉर्मडाटा () के साथ कई फ़ाइल अपलोड को कैसे संभाल सकता हूं?] इस उद्देश्य के लिए formData () विधि का उपयोग किया जा सकता है, जिससे आप एक ही अनुरोध में कई फाइलें भेज सकते हैं। document.getElementByid ('file...प्रोग्रामिंग 2025-04-06 पर पोस्ट किया गया
मैं फॉर्मडाटा () के साथ कई फ़ाइल अपलोड को कैसे संभाल सकता हूं?] इस उद्देश्य के लिए formData () विधि का उपयोग किया जा सकता है, जिससे आप एक ही अनुरोध में कई फाइलें भेज सकते हैं। document.getElementByid ('file...प्रोग्रामिंग 2025-04-06 पर पोस्ट किया गया -
 HTML स्वरूपण टैगHTML स्वरूपण तत्व ] HTML हमें CSS का उपयोग किए बिना पाठ को प्रारूपित करने की क्षमता प्रदान करता है। HTML में कई स्वरूपण टैग हैं। इन टैगों ...प्रोग्रामिंग 2025-04-06 पर पोस्ट किया गया
HTML स्वरूपण टैगHTML स्वरूपण तत्व ] HTML हमें CSS का उपयोग किए बिना पाठ को प्रारूपित करने की क्षमता प्रदान करता है। HTML में कई स्वरूपण टैग हैं। इन टैगों ...प्रोग्रामिंग 2025-04-06 पर पोस्ट किया गया -
 Chatbot कमांड निष्पादन के लिए वास्तविक समय में कैसे कैप्चर और स्ट्रीम करें?] हालाँकि, वास्तविक समय में स्टडआउट को पुनः प्राप्त करने का प्रयास करते समय चुनौतियां उत्पन्न होती हैं। इसे दूर करने के लिए, हमें स्क्रिप्ट के निष्पाद...प्रोग्रामिंग 2025-04-06 पर पोस्ट किया गया
Chatbot कमांड निष्पादन के लिए वास्तविक समय में कैसे कैप्चर और स्ट्रीम करें?] हालाँकि, वास्तविक समय में स्टडआउट को पुनः प्राप्त करने का प्रयास करते समय चुनौतियां उत्पन्न होती हैं। इसे दूर करने के लिए, हमें स्क्रिप्ट के निष्पाद...प्रोग्रामिंग 2025-04-06 पर पोस्ट किया गया -
 मान्य कोड के बावजूद PHP में इनपुट कैप्चरिंग इनपुट क्यों है?] $ _Server ['php_self']?> हालांकि, आउटपुट खाली रहता है। जबकि विधि = "प्राप्त करें" मूल रूप से काम करती है, विधि = "पोस्ट"...प्रोग्रामिंग 2025-04-06 पर पोस्ट किया गया
मान्य कोड के बावजूद PHP में इनपुट कैप्चरिंग इनपुट क्यों है?] $ _Server ['php_self']?> हालांकि, आउटपुट खाली रहता है। जबकि विधि = "प्राप्त करें" मूल रूप से काम करती है, विधि = "पोस्ट"...प्रोग्रामिंग 2025-04-06 पर पोस्ट किया गया -
 मैं पांडा डेटाफ्रेम में कुशलता से कॉलम का चयन कैसे करूं?] पंडों में, कॉलम का चयन करने के लिए विभिन्न विकल्प हैं। संख्यात्मक सूचकांक यदि कॉलम सूचकांक ज्ञात हैं, तो उन्हें चुनने के लिए ILOC फ़ंक्शन का ...प्रोग्रामिंग 2025-04-06 पर पोस्ट किया गया
मैं पांडा डेटाफ्रेम में कुशलता से कॉलम का चयन कैसे करूं?] पंडों में, कॉलम का चयन करने के लिए विभिन्न विकल्प हैं। संख्यात्मक सूचकांक यदि कॉलम सूचकांक ज्ञात हैं, तो उन्हें चुनने के लिए ILOC फ़ंक्शन का ...प्रोग्रामिंग 2025-04-06 पर पोस्ट किया गया -
 PHP में टाइमज़ोन को कुशलता से कैसे परिवर्तित करें?] यह गाइड अलग-अलग टाइमज़ोन के बीच तारीखों और समय को परिवर्तित करने के लिए एक आसान-से-प्रभाव विधि प्रदान करेगा। उदाहरण के लिए: // उपयोगकर्ता के Timez...प्रोग्रामिंग 2025-04-06 पर पोस्ट किया गया
PHP में टाइमज़ोन को कुशलता से कैसे परिवर्तित करें?] यह गाइड अलग-अलग टाइमज़ोन के बीच तारीखों और समय को परिवर्तित करने के लिए एक आसान-से-प्रभाव विधि प्रदान करेगा। उदाहरण के लिए: // उपयोगकर्ता के Timez...प्रोग्रामिंग 2025-04-06 पर पोस्ट किया गया -
 जावास्क्रिप्ट में कई चर घोषित करने के लिए कौन सी विधि अधिक बनाए रखने योग्य है?] इसके लिए दो सामान्य दृष्टिकोण हैं: प्रत्येक चर को एक अलग लाइन पर घोषित करना: var चर १ = "हैलो, दुनिया!" var चर 2 = "परीक्षण ...प्रोग्रामिंग 2025-04-06 पर पोस्ट किया गया
जावास्क्रिप्ट में कई चर घोषित करने के लिए कौन सी विधि अधिक बनाए रखने योग्य है?] इसके लिए दो सामान्य दृष्टिकोण हैं: प्रत्येक चर को एक अलग लाइन पर घोषित करना: var चर १ = "हैलो, दुनिया!" var चर 2 = "परीक्षण ...प्रोग्रामिंग 2025-04-06 पर पोस्ट किया गया
चीनी भाषा का अध्ययन करें
- 1 आप चीनी भाषा में "चलना" कैसे कहते हैं? #का चीनी उच्चारण, #का चीनी सीखना
- 2 आप चीनी भाषा में "विमान ले लो" कैसे कहते हैं? #का चीनी उच्चारण, #का चीनी सीखना
- 3 आप चीनी भाषा में "ट्रेन ले लो" कैसे कहते हैं? #का चीनी उच्चारण, #का चीनी सीखना
- 4 आप चीनी भाषा में "बस ले लो" कैसे कहते हैं? #का चीनी उच्चारण, #का चीनी सीखना
- 5 चीनी भाषा में ड्राइव को क्या कहते हैं? #का चीनी उच्चारण, #का चीनी सीखना
- 6 तैराकी को चीनी भाषा में क्या कहते हैं? #का चीनी उच्चारण, #का चीनी सीखना
- 7 आप चीनी भाषा में साइकिल चलाने को क्या कहते हैं? #का चीनी उच्चारण, #का चीनी सीखना
- 8 आप चीनी भाषा में नमस्ते कैसे कहते हैं? 你好चीनी उच्चारण, 你好चीनी सीखना
- 9 आप चीनी भाषा में धन्यवाद कैसे कहते हैं? 谢谢चीनी उच्चारण, 谢谢चीनी सीखना
- 10 How to say goodbye in Chinese? 再见Chinese pronunciation, 再见Chinese learning
























