 Page de garde > La programmation > Maîtriser la segmentation des images : comment les techniques traditionnelles brillent encore à l'ère numérique
Page de garde > La programmation > Maîtriser la segmentation des images : comment les techniques traditionnelles brillent encore à l'ère numérique
Maîtriser la segmentation des images : comment les techniques traditionnelles brillent encore à l'ère numérique
Introduction
La segmentation d'image, l'une des procédures les plus élémentaires de la vision par ordinateur, permet à un système de décomposer et d'analyser diverses régions d'une image. Qu'il s'agisse de reconnaissance d'objets, d'imagerie médicale ou de conduite autonome, la segmentation est ce qui décompose les images en parties significatives.
Bien que les modèles d'apprentissage profond continuent d'être de plus en plus populaires dans cette tâche, les techniques traditionnelles de traitement d'images numériques restent puissantes et pratiques. Les approches examinées dans cet article incluent le seuillage, la détection des contours, la région et le clustering en mettant en œuvre un ensemble de données bien reconnu pour l'analyse des images cellulaires, l'ensemble de données d'images MIVIA HEp-2.
Ensemble de données d'images MIVIA HEp-2
L'ensemble de données d'images MIVIA HEp-2 est un ensemble d'images des cellules utilisées pour analyser le profil des anticorps antinucléaires (ANA) à travers les cellules HEp-2. Il s’agit d’images 2D prises par microscopie à fluorescence. Cela le rend très adapté aux tâches de segmentation, notamment celles liées à l'analyse d'images médicales, où la détection de régions cellulaires est la plus importante.
Passons maintenant aux techniques de segmentation utilisées pour traiter ces images, en comparant leurs performances en fonction des scores F1.
1. Segmentation des seuils
Le seuillage est le processus par lequel les images en niveaux de gris sont converties en images binaires en fonction de l'intensité des pixels. Dans l’ensemble de données MIVIA HEp-2, ce processus est utile pour l’extraction de cellules en arrière-plan. Il est simple et efficace à un niveau relativement élevé, en particulier avec la méthode d'Otsu, car il calcule automatiquement le seuil optimal.
La méthode d'Otsu est une méthode de seuillage automatique, dans laquelle elle essaie de trouver la meilleure valeur de seuil pour produire la variance intra-classe minimale, séparant ainsi les deux classes : le premier plan (cellules) et l'arrière-plan. La méthode examine l'histogramme de l'image et calcule le seuil parfait, où la somme des variances d'intensité des pixels dans chaque classe est minimisée.
# Thresholding Segmentation
def thresholding(img):
# Convert image to grayscale
gray = cv.cvtColor(img, cv.COLOR_BGR2GRAY)
# Apply Otsu's thresholding
_, thresh = cv.threshold(gray, 0, 255, cv.THRESH_BINARY cv.THRESH_OTSU)
return thresh
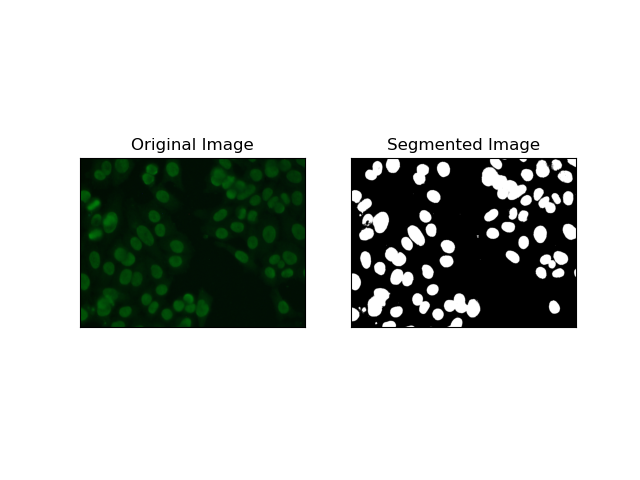
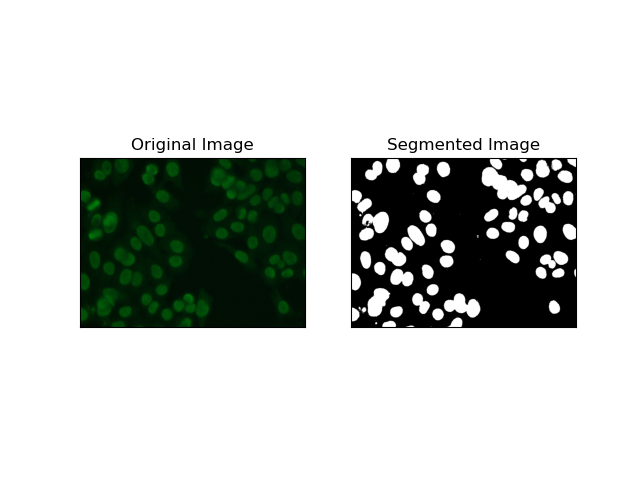
2. Segmentation de la détection des bords
La détection des contours concerne l'identification des limites d'objets ou de régions, telles que les contours des cellules dans l'ensemble de données MIVIA HEp-2. Parmi les nombreuses méthodes disponibles pour détecter les changements brusques d'intensité, le Canny Edge Detector est la méthode la meilleure et donc la plus appropriée à utiliser pour détecter les limites cellulaires.
Canny Edge Detector est un algorithme à plusieurs étapes qui peut détecter les bords en détectant les zones de forts gradients d'intensité. Le processus comprend le lissage avec un filtre gaussien, le calcul des gradients d'intensité, l'application d'une suppression non maximale pour éliminer les réponses parasites et une opération finale de double seuillage pour la conservation des seuls bords saillants.
# Edge Detection Segmentation
def edge_detection(img):
# Convert image to grayscale
gray = cv.cvtColor(img, cv.COLOR_BGR2GRAY)
# Apply Gaussian blur
gray = cv.GaussianBlur(gray, (3, 3), 0)
# Calculate lower and upper thresholds for Canny edge detection
sigma = 0.33
v = np.median(gray)
lower = int(max(0, (1.0 - sigma) * v))
upper = int(min(255, (1.0 sigma) * v))
# Apply Canny edge detection
edges = cv.Canny(gray, lower, upper)
# Dilate the edges to fill gaps
kernel = np.ones((5, 5), np.uint8)
dilated_edges = cv.dilate(edges, kernel, iterations=2)
# Clean the edges using morphological opening
cleaned_edges = cv.morphologyEx(dilated_edges, cv.MORPH_OPEN, kernel, iterations=1)
# Find connected components and filter out small components
num_labels, labels, stats, _ = cv.connectedComponentsWithStats(
cleaned_edges, connectivity=8
)
min_size = 500
filtered_mask = np.zeros_like(cleaned_edges)
for i in range(1, num_labels):
if stats[i, cv.CC_STAT_AREA] >= min_size:
filtered_mask[labels == i] = 255
# Find contours of the filtered mask
contours, _ = cv.findContours(
filtered_mask, cv.RETR_EXTERNAL, cv.CHAIN_APPROX_SIMPLE
)
# Create a filled mask using the contours
filled_mask = np.zeros_like(gray)
cv.drawContours(filled_mask, contours, -1, (255), thickness=cv.FILLED)
# Perform morphological closing to fill holes
final_filled_image = cv.morphologyEx(
filled_mask, cv.MORPH_CLOSE, kernel, iterations=2
)
# Dilate the final filled image to smooth the edges
final_filled_image = cv.dilate(final_filled_image, kernel, iterations=1)
return final_filled_image
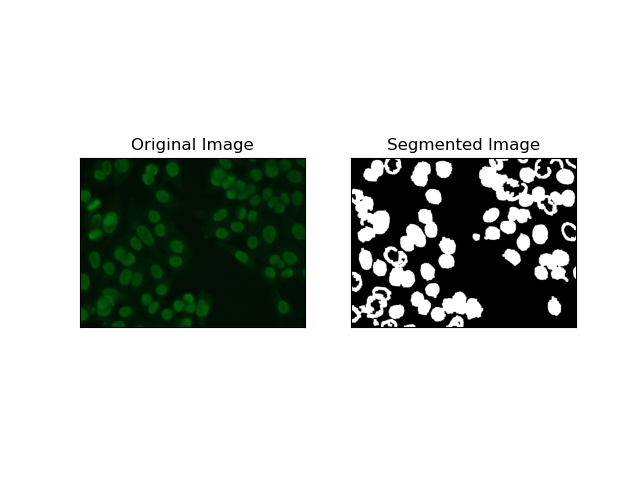
3. Segmentation par région
La segmentation basée sur les régions regroupe les pixels similaires en régions, en fonction de certains critères tels que l'intensité ou la couleur. La technique de Segmentation des bassins versants peut être utilisée pour aider à segmenter les images de cellules HEp-2 afin de pouvoir détecter les régions qui représentent les cellules ; il considère les intensités de pixels comme une surface topographique et décrit les régions distinctives.
LaLa segmentation des bassins versants traite les intensités des pixels comme une surface topographique. L'algorithme identifie des « bassins » dans lesquels il identifie des minima locaux puis inonde progressivement ces bassins pour agrandir des régions distinctes. Cette technique est très utile lorsqu'on veut séparer des objets en contact, comme dans le cas de cellules au sein d'images microscopiques, mais elle peut être sensible au bruit. Le processus peut être guidé par des marqueurs et la sur-segmentation peut souvent être réduite.
# Region-Based Segmentation
def region_based(img):
# Convert image to grayscale
gray = cv.cvtColor(img, cv.COLOR_BGR2GRAY)
# Apply Otsu's thresholding
_, thresh = cv.threshold(gray, 0, 255, cv.THRESH_BINARY_INV cv.THRESH_OTSU)
# Apply morphological opening to remove noise
kernel = np.ones((3, 3), np.uint8)
opening = cv.morphologyEx(thresh, cv.MORPH_OPEN, kernel, iterations=2)
# Dilate the opening to get the background
sure_bg = cv.dilate(opening, kernel, iterations=3)
# Calculate the distance transform
dist_transform = cv.distanceTransform(opening, cv.DIST_L2, 5)
# Threshold the distance transform to get the foreground
_, sure_fg = cv.threshold(dist_transform, 0.2 * dist_transform.max(), 255, 0)
sure_fg = np.uint8(sure_fg)
# Find the unknown region
unknown = cv.subtract(sure_bg, sure_fg)
# Label the markers for watershed algorithm
_, markers = cv.connectedComponents(sure_fg)
markers = markers 1
markers[unknown == 255] = 0
# Apply watershed algorithm
markers = cv.watershed(img, markers)
# Create a mask for the segmented region
mask = np.zeros_like(gray, dtype=np.uint8)
mask[markers == 1] = 255
return mask
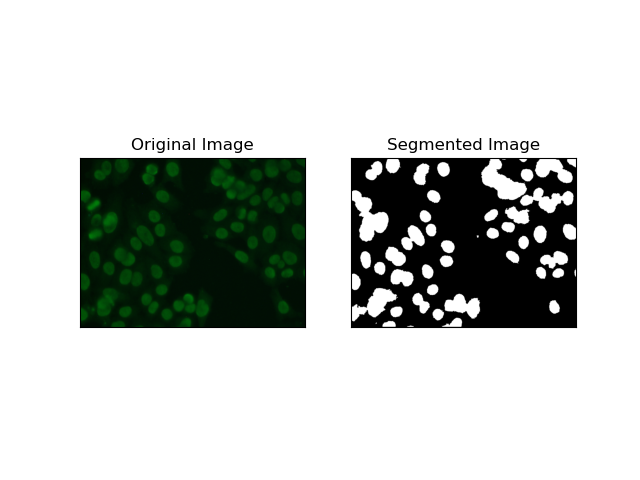
4. Segmentation basée sur le clustering
Les techniques de regroupement telles que K-Means ont tendance à regrouper les pixels en groupes similaires, ce qui fonctionne bien lorsque l'on souhaite segmenter des cellules dans des environnements multicolores ou complexes, comme le montrent les images de cellules HEp-2. Fondamentalement, cela pourrait représenter différentes classes, comme une région cellulaire par rapport à un arrière-plan.
K-means est un algorithme d'apprentissage non supervisé permettant de regrouper des images en fonction de la similarité des pixels en termes de couleur ou d'intensité. L'algorithme sélectionne de manière aléatoire K centroïdes, attribue chaque pixel au centroïde le plus proche et met à jour le centroïde de manière itérative jusqu'à ce qu'il converge. Il est particulièrement efficace pour segmenter une image comportant plusieurs régions d’intérêt très différentes les unes des autres.
# Clustering Segmentation
def clustering(img):
# Convert image to grayscale
gray = cv.cvtColor(img, cv.COLOR_BGR2GRAY)
# Reshape the image
Z = gray.reshape((-1, 3))
Z = np.float32(Z)
# Define the criteria for k-means clustering
criteria = (cv.TERM_CRITERIA_EPS cv.TERM_CRITERIA_MAX_ITER, 10, 1.0)
# Set the number of clusters
K = 2
# Perform k-means clustering
_, label, center = cv.kmeans(Z, K, None, criteria, 10, cv.KMEANS_RANDOM_CENTERS)
# Convert the center values to uint8
center = np.uint8(center)
# Reshape the result
res = center[label.flatten()]
res = res.reshape((gray.shape))
# Apply thresholding to the result
_, res = cv.threshold(res, 0, 255, cv.THRESH_BINARY cv.THRESH_OTSU)
return res
Évaluation des techniques à l'aide des scores F1
Le score F1 est une mesure qui combine précision et rappel pour comparer l'image de segmentation prédite avec l'image de vérité terrain. C'est le moyen harmonique de précision et de rappel, utile dans les cas de déséquilibre élevé des données, comme dans les ensembles de données d'imagerie médicale.
Nous avons calculé le score F1 pour chaque méthode de segmentation en aplatissant à la fois la vérité terrain et l'image segmentée et en calculant le score F1 pondéré.
def calculate_f1_score(ground_image, segmented_image):
ground_image = ground_image.flatten()
segmented_image = segmented_image.flatten()
return f1_score(ground_image, segmented_image, average="weighted")
Nous avons ensuite visualisé les scores F1 des différentes méthodes à l'aide d'un simple histogramme :
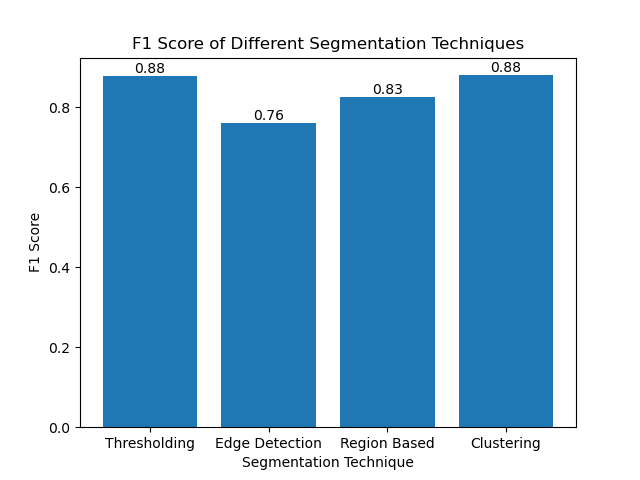
Conclusion
Bien que de nombreuses approches récentes de segmentation d'images émergent, les techniques de segmentation traditionnelles telles que le seuillage, la détection des contours, les méthodes basées sur les régions et le clustering peuvent être très utiles lorsqu'elles sont appliquées à des ensembles de données tels que l'ensemble de données d'images MIVIA HEp-2.
Chaque méthode a sa force :
- Le seuil est idéal pour une segmentation binaire simple.
- Edge Detection est une technique idéale pour la détection des limites.
- La segmentation Basée sur la région est très utile pour séparer les composants connectés de leurs voisins.
- Les méthodes de Clustering sont bien adaptées aux tâches de segmentation multirégionales.
En évaluant ces méthodes à l'aide des scores F1, nous comprenons les compromis que comporte chacun de ces modèles. Ces méthodes ne sont peut-être pas aussi sophistiquées que celles développées dans les modèles d'apprentissage profond les plus récents, mais elles restent rapides, interprétables et utilisables dans un large éventail d'applications.
Merci d'avoir lu ! J'espère que cette exploration des techniques traditionnelles de segmentation d'images inspirera votre prochain projet. N'hésitez pas à partager vos réflexions et expériences dans les commentaires ci-dessous !
-
 Pourquoi Java ne peut-il pas créer des tableaux génériques?Erreur de création de table ArrayList [2]; Java rapporte une erreur "création de tableau générique". Pourquoi cela n'est-il pas ...La programmation Publié le 2025-07-10
Pourquoi Java ne peut-il pas créer des tableaux génériques?Erreur de création de table ArrayList [2]; Java rapporte une erreur "création de tableau générique". Pourquoi cela n'est-il pas ...La programmation Publié le 2025-07-10 -
 Comment empêcher les soumissions en double après la rafraîchissement du formulaire?Empêcher les soumissions en double avec une manipulation de rafraîchissement dans le développement Web, il est courant d'informer le probl...La programmation Publié le 2025-07-10
Comment empêcher les soumissions en double après la rafraîchissement du formulaire?Empêcher les soumissions en double avec une manipulation de rafraîchissement dans le développement Web, il est courant d'informer le probl...La programmation Publié le 2025-07-10 -
 Comment combiner les données de trois tables MySQL dans un nouveau tableau?mysql: création d'un nouveau tableau à partir de données et de colonnes de trois tables Question: Comment puis-je créer un nouveau tab...La programmation Publié le 2025-07-10
Comment combiner les données de trois tables MySQL dans un nouveau tableau?mysql: création d'un nouveau tableau à partir de données et de colonnes de trois tables Question: Comment puis-je créer un nouveau tab...La programmation Publié le 2025-07-10 -
 Comment insérer efficacement les données dans plusieurs tables MySQL dans une seule transaction?insérer MySql dans plusieurs tables Tenter d'insérer des données dans plusieurs tables avec une seule requête MySQL peut donner des résult...La programmation Publié le 2025-07-10
Comment insérer efficacement les données dans plusieurs tables MySQL dans une seule transaction?insérer MySql dans plusieurs tables Tenter d'insérer des données dans plusieurs tables avec une seule requête MySQL peut donner des résult...La programmation Publié le 2025-07-10 -
 Comment gérer la saisie des utilisateurs dans le mode exclusif complet de Java?Gestion de la saisie de l'utilisateur en mode exclusif en plein écran en java introduction Lors de l'exécution d'une application...La programmation Publié le 2025-07-10
Comment gérer la saisie des utilisateurs dans le mode exclusif complet de Java?Gestion de la saisie de l'utilisateur en mode exclusif en plein écran en java introduction Lors de l'exécution d'une application...La programmation Publié le 2025-07-10 -
 Comment créer des variables dynamiques dans Python?Création de variables dynamiques dans python La capacité de créer des variables dynamiquement peut être un outil puissant, en particulier lors...La programmation Publié le 2025-07-10
Comment créer des variables dynamiques dans Python?Création de variables dynamiques dans python La capacité de créer des variables dynamiquement peut être un outil puissant, en particulier lors...La programmation Publié le 2025-07-10 -
 Comment télécharger des fichiers avec des paramètres supplémentaires à l'aide de java.net.urlconnection et de codage multipart / formulaire de formulaire?Téléchargement des fichiers avec des demandes http pour télécharger des fichiers sur un serveur http tout en soumettant des paramètres supplém...La programmation Publié le 2025-07-10
Comment télécharger des fichiers avec des paramètres supplémentaires à l'aide de java.net.urlconnection et de codage multipart / formulaire de formulaire?Téléchargement des fichiers avec des demandes http pour télécharger des fichiers sur un serveur http tout en soumettant des paramètres supplém...La programmation Publié le 2025-07-10 -
 Conseils pour trouver la position d'élément dans Java ArrayRécupération de la position de l'élément dans les tableaux java dans la classe des tableaux de Java, il n'y a pas de méthode directe &...La programmation Publié le 2025-07-10
Conseils pour trouver la position d'élément dans Java ArrayRécupération de la position de l'élément dans les tableaux java dans la classe des tableaux de Java, il n'y a pas de méthode directe &...La programmation Publié le 2025-07-10 -
 Comment récupérer la dernière bibliothèque jQuery des API Google?Récupération de la dernière bibliothèque jQuery à partir de Google API L'URL jQuery fournie dans la question est pour la version 1.2.6. Po...La programmation Publié le 2025-07-10
Comment récupérer la dernière bibliothèque jQuery des API Google?Récupération de la dernière bibliothèque jQuery à partir de Google API L'URL jQuery fournie dans la question est pour la version 1.2.6. Po...La programmation Publié le 2025-07-10 -
 Comment récupérer efficacement la dernière ligne pour chaque identifiant unique dans PostgreSQL?PostgreSQL: Extraction de la dernière ligne pour chaque identifiant unique Dans PostgreSql, vous pouvez rencontrer des situations de données o...La programmation Publié le 2025-07-10
Comment récupérer efficacement la dernière ligne pour chaque identifiant unique dans PostgreSQL?PostgreSQL: Extraction de la dernière ligne pour chaque identifiant unique Dans PostgreSql, vous pouvez rencontrer des situations de données o...La programmation Publié le 2025-07-10 -
 Comment résoudre les écarts de chemin du module dans GO Mod en utilisant la directive Remplacer?surmonter la divergence du chemin du module dans go mod Lors de l'utilisation de Go Mod, il est possible de rencontrer un conflit où un pa...La programmation Publié le 2025-07-10
Comment résoudre les écarts de chemin du module dans GO Mod en utilisant la directive Remplacer?surmonter la divergence du chemin du module dans go mod Lors de l'utilisation de Go Mod, il est possible de rencontrer un conflit où un pa...La programmation Publié le 2025-07-10 -
 Pourquoi est-ce que je reçois une erreur \ "class \ 'ziparchive \' non trouvée \" après avoir installé archive_zip sur mon serveur Linux?classe 'ziparchive' introuvable erreur lors de l'installation d'archive_zip sur le serveur Linux symptôme: Lorsque vous tent...La programmation Publié le 2025-07-10
Pourquoi est-ce que je reçois une erreur \ "class \ 'ziparchive \' non trouvée \" après avoir installé archive_zip sur mon serveur Linux?classe 'ziparchive' introuvable erreur lors de l'installation d'archive_zip sur le serveur Linux symptôme: Lorsque vous tent...La programmation Publié le 2025-07-10 -
 Analyse du langage fortement tapé CSSL'une des façons de classer un langage de programmation est de la force ou de la faiblesse. Ici, «tapé» signifie si les variables sont connues au...La programmation Publié le 2025-07-10
Analyse du langage fortement tapé CSSL'une des façons de classer un langage de programmation est de la force ou de la faiblesse. Ici, «tapé» signifie si les variables sont connues au...La programmation Publié le 2025-07-10 -
 Guide pour résoudre les problèmes CORS dans Spring Security 4.1 et plusSpring Security Cors Filter: dépannage des problèmes communs Lors de l'intégration de Spring Security dans un projet existant, vous pouvez...La programmation Publié le 2025-07-10
Guide pour résoudre les problèmes CORS dans Spring Security 4.1 et plusSpring Security Cors Filter: dépannage des problèmes communs Lors de l'intégration de Spring Security dans un projet existant, vous pouvez...La programmation Publié le 2025-07-10 -
 Pourquoi Pytz montre-t-il des décalages de fuseau horaire inattendus initialement?Dicontenance du fuseau horaire avec pytz Certains flammes de temps présentent des décalages particuliers lorsqu'ils sont initialement obte...La programmation Publié le 2025-07-10
Pourquoi Pytz montre-t-il des décalages de fuseau horaire inattendus initialement?Dicontenance du fuseau horaire avec pytz Certains flammes de temps présentent des décalages particuliers lorsqu'ils sont initialement obte...La programmation Publié le 2025-07-10
Étudier le chinois
- 1 Comment dit-on « marcher » en chinois ? 走路 Prononciation chinoise, 走路 Apprentissage du chinois
- 2 Comment dit-on « prendre l’avion » en chinois ? 坐飞机 Prononciation chinoise, 坐飞机 Apprentissage du chinois
- 3 Comment dit-on « prendre un train » en chinois ? 坐火车 Prononciation chinoise, 坐火车 Apprentissage du chinois
- 4 Comment dit-on « prendre un bus » en chinois ? 坐车 Prononciation chinoise, 坐车 Apprentissage du chinois
- 5 Comment dire conduire en chinois? 开车 Prononciation chinoise, 开车 Apprentissage du chinois
- 6 Comment dit-on nager en chinois ? 游泳 Prononciation chinoise, 游泳 Apprentissage du chinois
- 7 Comment dit-on faire du vélo en chinois ? 骑自行车 Prononciation chinoise, 骑自行车 Apprentissage du chinois
- 8 Comment dit-on bonjour en chinois ? 你好Prononciation chinoise, 你好Apprentissage du chinois
- 9 Comment dit-on merci en chinois ? 谢谢Prononciation chinoise, 谢谢Apprentissage du chinois
- 10 How to say goodbye in Chinese? 再见Chinese pronunciation, 再见Chinese learning
























